【佳学基因检测】多发性硬化症基因检测
基因检测导读:
多发性硬化症(MS)是一种中枢神经系统自身免疫性疾病。根据《多发性硬化症基因检测数据集》,中国约有160万到400万人受此疾病困扰,全球范围内约有800万人受此疾病影响。女性患MS的可能性是男性的两倍,部分原因是两性之间激素和遗传对免疫系统的影响存在差异。激活的T细胞淋巴细胞介导的炎症通常会导致神经元轴突脱髓鞘,从而引发MS的神经症状,并导致脑损伤。由于MS的早期症状往往轻微且多变,这些症状由环境触发的CD4+自身反应性T细胞引起,且需要数年时间才能发展成明显的神经系统表型的慢性疾病状态,因此早期识别MS非常困难。尽管先进的成像技术和脑脊液/血液生物标志物可以检测到MS的进展,这些策略通常需要初始临床事件作为触发条件,从而限制了早期检测的可能性。如果MS没有得到及时治疗,神经损伤将变得不可逆转。如果能够在早期阶段发现MS,许多新疗法在临床应用方面已有显著进展。因此,发现新的风险标志物和评估MS的相关知识对于早期检测至关重要。
初步研究表明,MS存在非环境性的多基因遗传风险。全基因组关联研究(GWAS)彻底改变了多发性硬化症基因检测项目组对免疫系统在疾病中作用的认识;然而,这些研究仅解释了约四分之一的遗传风险。来自MS遗传学双胞胎研究的遗传力估计表明,同卵双胞胎的一致率为25-30%,而异卵双胞胎的一致率较低,这表明多种遗传因素和环境影响对疾病发展存在复杂的相互作用。为了更好地理解这些罕见的多基因对MS的影响,全基因组/外显子组测序已开始识别导致疾病的罕见变异。基于这些遗传学见解构建的基因网络将有助于建立系统风险评估,同时在个性化医疗策略中扩展发现其他导致MS的罕见遗传变异的途径。
通过发现MS发生和发展的新机制,基因检测有可能开发出新的检测方法和靶向治疗。这一点尤为重要,因为多发性硬化症基因检测项目组知道GWAS队列缺乏种族多样性,这些研究中使用的多重假设检验的多样性不足。多发性硬化症基因检测项目组之前已展示了解决GWAS中罕见变异的基因组机制的能力,这些变异因种族多样性而缺乏动力,因此多发性硬化症基因检测项目组提出了同样的系统生物学策略来研究MS病因,以进一步了解罕见和常见的共同遗传对疾病的贡献。
多发性硬化症致病基因鉴定基因解码中提出的流程提供了一种利用与MS相关的GWAS单核苷酸多态性(SNP)的方法,通过研究连锁不平衡(LD)SNP的机制来发现途径和交叉兴趣点。研究了LD基因突变序列及之前与MS相关的研究中的基因,以确定有害的编码序列基因突变序列及其对基因调控的影响(图1)。因此,多发性硬化症致病基因鉴定基因解码为复杂剖析常见和罕见变异及其之间的系统通路相互作用提供了一个框架,这些相互作用导致了MS的多基因特性。
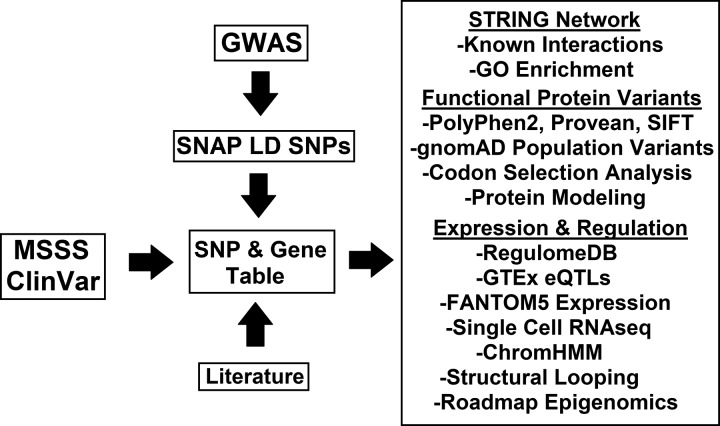
图1:用于识别和研究与多发性硬化症有关的基因/变异的工作流程。
如何进行多发性硬化症的致病基因鉴定基因解码?
找出多化性硬化病的致病基因和突变位点
对于所有提及“多发性硬化症”的性状,多发性硬化症基因检测项目组从 EBI/NHGRI GWAS 目录 中提取了主要 SNP。然后使用 SNAP和 0.8 相关性截止值分析所有 LD SNP 的主要 SNP,并删除冗余基因突变序列。来自多发性硬化症严重程度评分 (MSSS) 关联 ) 的基因和来自 ClinVar的“多发性硬化症”下列出的所有基因/基因突变序列均被纳入列表。然后使用 STRING对所有基因进行评估,使用基因本体 (GO) 富集来分析分子功能、KEGG 通路和 PMID 基因关联。
蛋白质结构与功能变化分析
使用PolyPhen2 、SIFT 和 Provean工具对来自 ClinVar 和 gnomAD 的所有变异进行错义功能评估,评估每个具有错义变异的基因。至少被其中一种工具鉴定为有功能的变异被纳入多发性硬化症基因检测项目组的候选基因列表 (表格1) 并进行多发性硬化症基因检测项目组之前描述的深度序列-结构-功能分析。通过 I-TASSER为细胞内和细胞外结构域生成 CD6 蛋白模型,然后设计跨膜螺旋、合并模型,并使用 YASARA 将蛋白质嵌入 PEA 跨膜中。使用指定为 1 的每个工具的破坏性调用,然后按照以下方式生成每个错义基因突变序列的基因突变序列分数: (PolyPhen2 + SIFT + Proven + 保守性分数 ) × (21 个密码子线性基序保守性)。对于保守性分数为 1 或更高的基因突变序列,将来自 gnomAD 的等位基因计数乘以上述分数以计算群体影响。
表1:根据 GWAS、eQTL、ClinVar 和文献,查找多发性硬化症的主要基因
| 基因 | 纳入理由 | 候选位点 | 先导位点SNP | 与先导SNP 的距离 | R2 | 特征 | P 值 | OR | 参考文献 | 权重 |
| CD6† | PolyPhen2/SIFT | JX112MS30562 | rs17824933 | 15574 | 0.943 | 多发性硬化症 | 4.00E-09 | 1.18 | 19525953 | 高的 |
| CD6† | Provean/SIFT | JX207MS4233 | rs17824933 | 24852 | 0.836 | 多发性硬化症 | 4.00E-09 | 1.18 | 19525953 | 高的 |
| EVI5† | Provean/SIFT | JX118MS8092 | rs11808092 | 0 | 1 | EB 病毒核抗原 1 IgG 水平或多发性硬化症 | 2.00E-08 | 0 | 26819262 | 高的 |
| DKKL1† | Provean/SIFT | JX230MS3759 | rs2303759 | 0 | 1 | 多发性硬化症 | 5.00E-09 | 1.11 | 21833088 | 高的 |
| HNRNPA1† | 临床研究 | JXMS | — | — | — | — | — | — | — | 高的 |
| NR1H3† | 临床研究 | JXMS | — | — | — | — | — | — | — | 高的 |
| FAM69A | 胫神经基因表达谱 | JXMS | rs6604026 | — | — | 多发性硬化症 | 8.00E-06 | 1.15 | 17660530 | 高的 |
| HLA-DRB1* | 胫神经基因表达谱 | JX927MS980 | rs9271640 | 18291 | 0.861 | 多发性硬化症中的寡克隆带状态 | 5.00E-12 | 1.56 | 25616667 | 高的 |
| HLA-DRB5* | 胫神经 / 脾脏 eQTL | JXMS | rs9271640 | — | — | 多发性硬化症中的寡克隆带状态 | 5.00E-12 | 1.56 | 25616667 | 高的 |
| KIF1B‡ | 胫神经基因表达谱 | JX十MS | rs10492972 | — | — | 多发性硬化症 | 3.00E-10 | 1.34 | 18997785 | 高的 |
| LINC00886 | 胫神经 / 脾脏 eQTL | JX867MS436 | rs12638253 | 91510 | 0.846 | 多发性硬化症(严重程度) | 2.00E-06 | 0 | 19010793 | 高的 |
| METTL15P1 | 胫神经基因表达谱 | JXMS | rs12638253 | — | — | 多发性硬化症(严重程度) | 2.00E-06 | 0 | 19010793 | 高的 |
| NAP1L4P1 | 胫神经基因表达谱 | JX十五MS | rs12025416 | — | — | 多发性硬化症 | 1.00E-07 | 1.45 | 21244703 | 高的 |
| PCDHGC3 | 胫神经基因表达谱 | JX十九MS | rs1062158 | — | — | 多发性硬化症 | 2.00E-06 | 1.08 | 21833088 | 高的 |
| RGS14 | 胫神经基因表达谱 | JXMS | rs4075958 | — | — | 多发性硬化症 | 5.00E-07 | 1.09 | 21833088 | 高的 |
| RP11–10L12.2 | 胫神经基因表达谱 | JX十九MS | rs228614 | — | — | 多发性硬化症 | 1.00E-07 | 1.09 | 21833088 | 高的 |
| RPL37 | 胫神经基因表达谱 | JX十七MS | rs6896969 | — | — | 多发性硬化症 | 2.00E-07 | 1.1 | 19525953 | 高的 |
| TIPARP | 胫神经基因表达谱 | JX289MS3226 | rs12638253 | 101871 | 0.875 | 多发性硬化症(严重程度) | 2.00E-06 | 0 | 19010793 | 高的 |
| TMEM87B | 胫神经基因表达谱 | JX689MS6969 | rs17174870 | 148190 | 0.821 | 多发性硬化症 | 1.00E-08 | 1.11 | 21833088 | 高的 |
| TRIM2 | 胫神经基因表达谱 | JX687MS9489 | rs12644284 | 0 | 1 | 多发性硬化症 | 4.00E-06 | 2.04 | 21654844 | 高的 |
| FAM213B | 全血eQTL | JX十八MS | rs4648356 | — | — | 多发性硬化症 | 1.00E-14 | 1.14 | 21833088 | 高的 |
| FCRL3 | 全血/脾脏 eQTL | JXMS | rs3761959 | — | — | 多发性硬化症 | 3.00E-06 | 1.08 | 21833088 | 高的 |
| MERTK | 全血eQTL | JXMS | rs17174870 | — | — | 多发性硬化症 | 1.00E-08 | 1.11 | 21833088 | 高的 |
| MMEL1 | 全血/脾脏 eQTL | JXMS | rs4648356 | — | — | 多发性硬化症 | 1.00E-14 | 1.14 | 21833088 | 高的 |
| AHI1 | 脾脏基因座 | JXMS | rs11154801 | — | — | 多发性硬化症 | 1.00E-13 | 1.13 | 21833088 | 高的 |
| TBKBP1 | 顺式eQTL 1b | JX479MS4048 | rs8070463 | 5763 | 0.837 | 多发性硬化症 | 1.00E-07 | 1.15 | 22190364 | 高的 |
| DEXI | 顺式eQTL 1b | JX649MS8169 | rs6498169 | 0 | 1 | 多发性硬化症 | 4.00E-06 | 1.14 | 17660530 | 高的 |
| SEZ6L2 | 顺式eQTL 1b | JX102MS4 | rs34286592 | 6058 | 0.929 | 多发性硬化症 | 5.00E-07 | 1.16 | 27386562 | 高的 |
| PGD | 顺式eQTL 1b | JX373MS7155 | rs10492972 | 106667 | 1 | 多发性硬化症 | 3.00E-10 | 1.34 | 18997785 | 高的 |
| CLTB | 顺式eQTL 1b | JX117MS46443 | rs4075958 | 13794 | 0.927 | 多发性硬化症 | 5.00E-07 | 1.09 | 21833088 | 高的 |
| RMND5B | 顺式eQTL 1b | JX117MS46443 | rs4075958 | 13794 | 0.927 | 多发性硬化症 | 5.00E-07 | 1.09 | 21833088 | 高的 |
| TMED9 | 顺式eQTL 1b | JX117MS46443 | rs4075958 | 13794 | 0.927 | 多发性硬化症 | 5.00E-07 | 1.09 | 21833088 | 高的 |
| LRRC34 | PolyPhen | JX109MS36600 | rs10936599 | 22484 | 0.956 | 多发性硬化症 | 7.00E-07 | 1.1 | 21833088 | 中等的 |
| MPV17L2 | Provean | JX874MS628 | rs874628 | 0 | 1 | 多发性硬化症 | 1.00E-08 | 1.11 | 21833088 | 中等的 |
| IKZF2 | RegDB 1b TF | JX479MS4048 | rs8070463 | 5763 | 0.837 | 多发性硬化症 | 1.00E-07 | 1.15 | 22190364 | 中等的 |
| HES1 | RegDB 1b TF | JX649MS8169 | rs6498169 | 0 | 1 | 多发性硬化症 | 4.00E-06 | 1.14 | 17660530 | 中等的 |
| TGIF1 | RegDB 1b TF | JX102MS4 | rs34286592 | 6058 | 0.929 | 多发性硬化症 | 5.00E-07 | 1.16 | 27386562 | 中等的 |
| RGS1 | 顺式eQTL 1d | JX281MS6305 | rs1323292 | 16752 | 1 | 多发性硬化症 | 2.00E-08 | 1.12 | 21833088 | 中等的 |
| CDK2AP1 | 顺式eQTL 1d | JX655MS293 | rs1790100 | 128320 | 0.894 | 多发性硬化症 | 7.00E-07 | 1.11 | 19525953 | 中等的 |
| CD40 | 顺式eQTL 1d | JX242MS5752 | rs2425752 | 0 | 1 | 多发性硬化症 | 5.00E-10 | 1.11 | 21833088 | 中等的 |
| MVP | 顺式eQTL 1d | JX993MS8630 | rs34286592 | 16628 | 1 | 多发性硬化症 | 5.00E-07 | 1.16 | 27386562 | 中等的 |
| PRRT2 | 顺式eQTL 1d | JX993MS8630 | rs34286592 | 16628 | 1 | 多发性硬化症 | 5.00E-07 | 1.16 | 27386562 | 中等的 |
| CDC37 | 顺式eQTL 1d | JX129MS78984 | rs8112449 | 827 | 1 | 多发性硬化症 | 1.00E-06 | 1.08 | 21833088 | 中等的 |
| GABPA | RegDB 2a TF | JX562MS35845 | rs4075958 | 13528 | 0.964 | 多发性硬化症 | 5.00E-07 | 1.09 | 21833088 | 中等的 |
| CTCF | RegDB 2a TF | JX798MS6874 | rs17594362 | 10364 | 0.887 | 多发性硬化症 | 4.00E-06 | 1.11 | 21833088 | 中等的 |
| EGR1 | RegDB 2a TF | JX128MS89006 | rs4902647 | 6372 | 0.967 | 多发性硬化症 | 9.00E-12 | 1.11 | 21833088 | 中等的 |
| YY1 | RegDB 2a TF | JX118MS78602 | rs10411936 | 6778 | 1 | 多发性硬化症 | 2.00E-07 | 1.16 | 22190364 | 中等的 |
| GFI1‡ | RegDB 2a TF | JX617MS84580 | rs10492972 | 82616 | 0.802 | 多发性硬化症 | 3.00E-10 | 1.34 | 18997785 | 中等的 |
| SPI1 | RegDB 2a TF | JX298MS4920 | rs1323292 | 3774 | 1 | 多发性硬化症 | 2.00E-08 | 1.12 | 21833088 | 中等的 |
| CLOCK | RegDB 2a TF | JX126MS24433 | rs2425752 | 21267 | 1 | 多发性硬化症 | 5.00E-10 | 1.11 | 21833088 | 中等的 |
| ARNTL | RegDB 2a TF | JX126MS24433 | rs2425752 | 21267 | 1 | 多发性硬化症 | 5.00E-10 | 1.11 | 21833088 | 中等的 |
| BACH1 | RegDB 2a TF | JX791MS40028 | rs6984045 | 284668 | 1 | 多发性硬化症 | 2.00E-06 | 1.59 | 19525955 | 中等的 |
| ASAP1 | RegDB 2a 基因内含子中的 SNP | JX791MS40028 | rs6984045 | 284668 | 1 | 多发性硬化症 | 2.00E-06 | 1.59 | 19525955 | 低的 |
| CDIPT-AS1 | RegDB 1b 基因内含子中的 SNP | JX102MS4 | rs34286592 | 6058 | 0.929 | 多发性硬化症 | 5.00E-07 | 1.16 | 27386562 | 低的 |
| CLEC16A | RegDB 1b 基因内含子中的 SNP | JX649MS8169 | rs6498169 | 0 | 1 | 多发性硬化症 | 4.00E-06 | 1.14 | 17660530 | 低的 |
| CLECL1 | RegDB 2a 基因内含子中的 SNP | JX104MS92165 | rs10466829 | 9870 | 0.935 | 多发性硬化症 | 1.00E-08 | 1.09 | 21833088 | 低的 |
| EPS15L1 | RegDB 2a 基因内含子中的 SNP | JX118MS78602 | rs10411936 | 6778 | 1 | 多发性硬化症 | 2.00E-07 | 1.16 | 22190364 | 低的 |
| KPNB1 | RegDB 1b 基因内含子中的 SNP | JX479MS4048 | rs8070463 | 5763 | 0.837 | 多发性硬化症 | 1.00E-07 | 1.15 | 22190364 | 低的 |
| SLC12A5 | RegDB 2a 基因内含子中的 SNP | JX126MS24433 | rs2425752 | 21267 | 1 | 多发性硬化症 | 5.00E-10 | 1.11 | 21833088 | 低的 |
| AFF3 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs12471490 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 3.15 × 10−5 | 0.56 | 21654844 | 低的 |
| ANAPC1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs4848821 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 1.44 × 10−5 | 0.8 | 21654844 | 低的 |
| ASXL2 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs10178552 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 4.76 × 10−5 | −0.41 | 21654844 | 低的 |
| CDH13 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs8047176 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 6.90 × 10−5 | 1.58 | 21654844 | 低的 |
| CDHR3 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs193806 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 5.53 × 10−5 | 0.63 | 21654844 | 低的 |
| CRTAC1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs11189446 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.05 × 10−5 | 0.54 | 21654844 | 低的 |
| CTNND2 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs11750073 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 6.49 × 10−5 | 1.99 | 21654844 | 低的 |
| FHIT | 多发性硬化症严重程度评分(MSSS) | JXMS | rs1735457 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 1.55 × 10−5 | 2.85 | 21654844 | 低的 |
| GPC5 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs17430373 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 3.12 × 10−5 | 4.19 | 21654844 | 低的 |
| GPR158 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7071606 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 4.25 × 10−5 | 3.59 | 21654844 | 低的 |
| GRIN2A | 多发性硬化症严重程度评分(MSSS) | JXMS | rs1448239 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.99 × 10−5 | 1.65 | 21654844 | 低的 |
| HACE1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7741733 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 1.97 × 10−5 | 1.69 | 21654844 | 低的 |
| IFNA10 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs10811505 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.56 × 10−5 | −0.50 | 21654844 | 低的 |
| IFNA16 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs1820314 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.13 × 10−5 | −0.50 | 21654844 | 低的 |
| IFNA17 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs9298814 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.30 × 10−5 | −0.50 | 21654844 | 低的 |
| JAZF1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs735664 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 5.97 × 10−5 | 1.65 | 21654844 | 低的 |
| KCNMA1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7087337 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 7.47 × 10−5 | 2 | 21654844 | 低的 |
| KLHL9 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs8729 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 3.85 × 10−5 | −0.49 | 21654844 | 低的 |
| LOC100289506 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7789940 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 6.04 × 10−6 | 1.87 | 21654844 | 低的 |
| MICB | 多发性硬化症严重程度评分(MSSS) | JXMS | rs2855814 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.09 × 10−5 | 0.4 | 21654844 | 低的 |
| NKD1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs12596811 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 1.80 × 10−5 | 2.43 | 21654844 | 低的 |
| NOS1AP | 多发性硬化症严重程度评分(MSSS) | JXMS | rs12403202 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 7.83 × 10−5 | 0.63 | 21654844 | 低的 |
| NPSR1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs17170015 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 4.65 × 10−5 | −0.48 | 21654844 | 低的 |
| OR6T1 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs1476203 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 4.43 × 10−5 | 0.65 | 21654844 | 低的 |
| OR8D4 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7942047 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 8.68 × 10−5 | 0.67 | 21654844 | 低的 |
| PDZD2 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs161522 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 4.35 × 10−5 | 1.62 | 21654844 | 低的 |
| PLCG2 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7185362 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 4.48 × 10−5 | 1.7 | 21654844 | 低的 |
| POPDC3 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs11962089 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 8.33 × 10−6 | −0.69 | 21654844 | 低的 |
| PPARGC1A | 多发性硬化症严重程度评分(MSSS) | JXMS | rs8192678 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 5.94 × 10−5 | 1.72 | 21654844 | 低的 |
| PRDM2 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs4344326 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 5.82 × 10−5 | 4.26 | 21654844 | 低的 |
| PSD3 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7015570 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 3.76 × 10−5 | −0.60 | 21654844 | 低的 |
| PTPRD | 多发性硬化症严重程度评分(MSSS) | JXMS | rs10977017 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 1.02 × 10−5 | 1.67 | 21654844 | 低的 |
| RBM20 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs1832745 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.02 × 10−5 | −0.45 | 21654844 | 低的 |
| RELN | 多发性硬化症严重程度评分(MSSS) | JXMS | rs10487166 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 5.33 × 10−5 | 0.45 | 21654844 | 低的 |
| STX8 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs7219526 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 7.14 × 10−5 | 0.64 | 21654844 | 低的 |
| YWHAG | 多发性硬化症严重程度评分(MSSS) | JXMS | rs17149161 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 5.83 × 10−6 | 1.87 | 21654844 | 低的 |
| ZFPM2 | 多发性硬化症严重程度评分(MSSS) | JXMS | rs10505082 | — | — | 多发性硬化症严重程度评分(MSSS) 协会 | 2.45 × 10−5 | 0.62 | 21654844 | 低的 |
†基因用匕首标记的位点有额外的蛋白质影响分析图 3。
*在 eQTL 分析中更详细地细分图 5。
‡单个 LD 块 (rs10492972) 中描述的多个数据集的连接图 6。
基因表达
每种基因的表达数据均取自 FANTOM或小鼠单细胞数据库。使用 Morpheus 在线工具可视化表达,并使用以下公式将表达水平转换为 z 分数:(样本值 - 所有组织中基因的平均值)/ 所有组织中基因值的标准差。对于单细胞分析,确定表达的基因是基于计数 >10 的基因。计数 >10 的细胞按所有其他基因聚类,并与计数 <10 的细胞进行比较,计算表达水平的 log2 倍数变化和计数 >10 的细胞百分比。
非编码基因突变序列基因突变
首先通过 RegulomeDB 工具评估所有 LD SNP 的 GWAS多发性硬化症(MS)相关区域,整理出发生改变的转录因子 (TF) 结合位点以及 TF 结合附近的基因。使用 hg38 注释整理出在 LD 区块 ± 50,000 kB 内发现的完整基因列表。从 GTEx 中提取表达数量性状基因座 (eQTL) 数据,并将其标准化为具有 eQTL 的组织内总基因数量(具有 eQTL 的多发性硬化症(MS)基因百分比/[组织 eQTL/所有组织中的平均 eQTL]),并查询 LD 区块基因以查找在胫神经、全血或脾脏数据集中发现的任何作为 eQTL 的 SNP。 LD区块注释碱基的 ChromHMM 模型来自 Roadmap Epigenomics。然后使用 SNAP(截止值为 0.8 R 2)重新评估顶级功能注释 SNP 的 LD,随后提取 GWAS 数据库中注释的所有 LD SNP,以确定 LD 区块的其他特征关联。
对基因进行排序
高优先级基因被注释为在 2/3 种工具(PolyPhen2、Provean 或 SIFT)中具有功能预测结果的错义变异、在 ClinVar 中具有多个映射变异或具有基因 eQTL 调控的 GWAS 基因座。中等优先级基因是在一种工具中具有功能预测结果的错义变异或 RegulomeDB 优先考虑的 TF 结合变异。低优先级基因是定位在功能性 TF 结合位点附近而没有 eQTL 的基因或通过 多发性硬化症严重程度评分(MSSS) 关联识别的不属于上述两组的基因。
多发性硬化症的发生如何受基因的影响?
MS 基因列表
从 GWAS 数据库中,有 218 个主要 SNP 与以下一项或多项相关:Epstein-Barr 病毒核抗原 1 IgG 水平或多发性硬化症、多发性硬化症中的免疫球蛋白 G 指数水平、多发性硬化症、多发性硬化症(发病年龄)、多发性硬化症(OCB 状态)、多发性硬化症(严重程度)、多发性硬化症或肌萎缩侧索硬化症、多发性硬化症-脑谷氨酸水平、多发性硬化症中的寡克隆带状态、多发性硬化症中的复发以及多发性硬化症对干扰素 β 的反应。从这些主要 SNP 中,有 3,505 个 SNP 处于 LD 中。其中 8 个被识别为 RegulomeDB 评分为 1b(eQTL 和 TF 结合预测变化,位点处已知多个 ENCODE TF),12 个被识别为 2a(TF 结合预测变化,位点处已知多个 ENCODE TF,包括与改变的结合位点匹配的 ENCODE),这表明在 LD 块内,多个 SNP 可能会改变基因调控。
根据上文讨论的多发性硬化症(MS)文献、ClinVar 和 GWAS 相关基因位点图谱对基因进行分析,得到了 96 个非重复的候选基因(表1)。其中,32 个为高优先级(33%),20 个为中等优先级(21%),44 个为低优先级(46%)。在 43 个具有 R 2数据的预测功能性 SNP 中,11 个的值范围为 0.802 至 0.894(26%,0.8 为 LD 映射的截止值),11 个的范围为 0.927–0.967(26%),20 个的值为 1(47%,与人类群体完全相关)。
已识别基因的多发性硬化症(MS)通路
开始了解基因的通路和网络表1,多发性硬化症基因检测项目组对 96 个基因使用了 STRING 工具,返回了 89 个映射基因/节点(图 2)。网络中的连接是实验确定的相互作用(19 个连接)、共表达(22 个连接)、注释数据库(22 个连接)和文本挖掘(42 个连接)。蛋白质-蛋白质相互作用的数量显著丰富于随机预期(P值 2e-7),表明基因列表中存在重叠的生物学途径。这突出表明,在表1并不是随机的,而是通过重叠的生物机制和途径聚集在一起的。
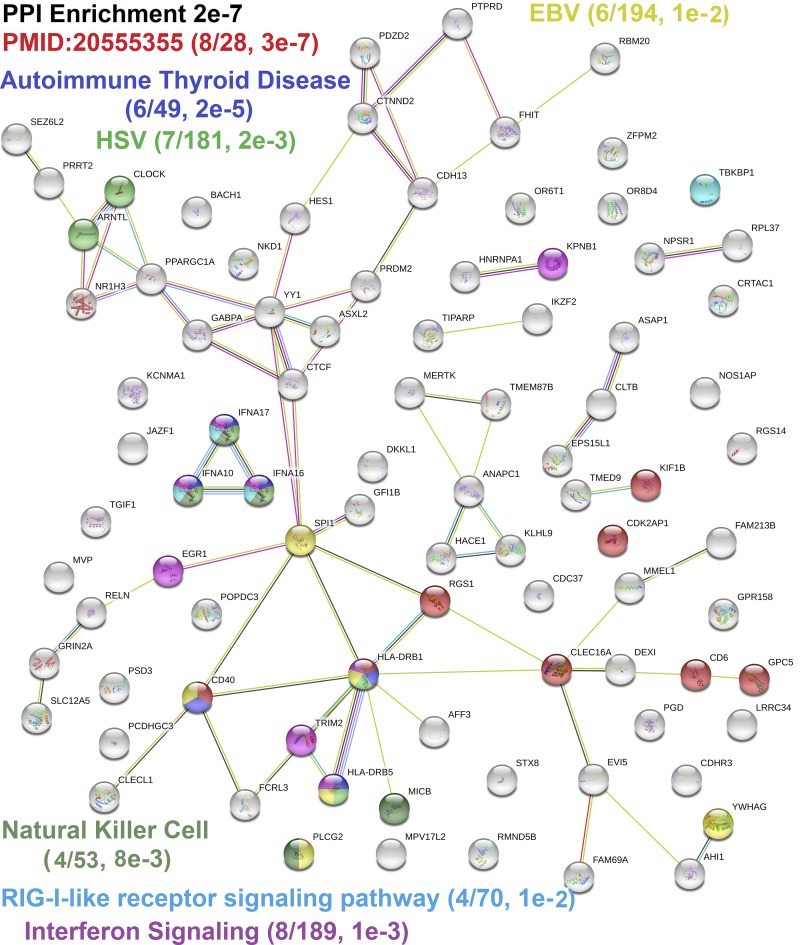
图 2:STRING 网络和基因本体论 (GO) 多发性硬化症基因富集。使用以下基因创建网络表1. 显著富集的 GO 术语显示为在整个基因组中与术语总数映射的基因数量以及每个基因的错误发现率 (FDR)。
在 89 个已映射基因列表中显著富集的 GO 术语网络包括:11 个(12.4%)为 TF(错误发现率,FDR 为 4e-4),6 个(6.7%)与自身免疫性甲状腺疾病(FDR 2e-05)相关,4 个(4.5%)与 RIG-1 样受体信号传导(FDR 1e-2)相关,4 个(4.5%)与自然杀伤细胞活化(FDR 8e-3)相关。此外,单纯疱疹感染(7 个基因,FDR 2e-3)和 Epstein-Barr 病毒感染(6 个基因,FDR 1e-2)基因也有富集,此前有研究表明这些基因与多发性硬化症(MS)有关。将这些结果与文献进行关联,可证实自身免疫性甲状腺疾病与多发性硬化症(MS)患者及其一级亲属共病;表达 NKG2D 受体的自然杀伤细胞可能对表达 NKG2D 配体的成体少突胶质细胞和胎儿星形胶质细胞具有细胞毒性,从而导致多发性硬化症(MS)中的病变;RIG-1 样受体通路的 TBKBP1 在脂肪细胞和神经元中高表达,且多发性硬化症(MS)中的抗 DNA 抗体升高。总体而言,这表明基于网络分析,MS 基因网络丰富了多层次的免疫和病毒反应生物学。
错义基因突变序列的分析
对于 GWAS,错义变异最容易进行功能分析,因此也是多发性硬化症基因检测项目组了解多发性硬化症(MS)遗传机制的起点。从 GWAS 的 LD SNP 中,多发性硬化症基因检测项目组在 12 种蛋白质(CD6、CLEC2D、DKKL1、EVI5、FCRL3、IL7R、LRRC34、MANBA、MMEL1、MPV17L2、SCO2、TNFRSF1A)中发现了 15 个错义 SNP。在 15 个错义变异中,有 8 个在 PolyPhen2/Provean/SIFT 中被预测为良性,一个在 PolyPhen 2 中可能是有害的,一个在 PolyPhen2 中可能是有害的而在 SIFT 中是有害的,一个在 Provean 中是有害的,一个在 PolyPhen2 中可能是有害的而在 SIFT 中是有害的,三个在 Provean 中是有害的而在 SIFT 中是有害的(表 2)。此外,ClinVar 中的两种蛋白质具有与多发性硬化症(MS)相关的错义基因突变序列,即 NR1H3 和 HNRNPA1。多发性硬化症基因检测项目组使用多发性硬化症基因检测项目组的基因突变序列分析工具分析了 GWAS 中的三种蛋白质(CD6、EVI5、DKKL1),其中至少有两种工具报告了破坏性预测,以及 ClinVar 中的两种蛋白质(NR1H3、HNRNPA1)(图 3,A – C)。
表 2.错义变异分析
| 基因(基因突变序列) | PolyPhen2 | Provean | SIFT |
|---|---|---|---|
| CD6 (T217M) | 大概 | 中性的 | 破坏 |
| EVI5 (Q612H) | 良性 | 有害的 | 破坏 |
| CD6 (G606S) | 良性 | 有害的 | 破坏 |
| DKKL1(M109R) | 良性 | 有害的 | 破坏 |
| LRRC34(L241I) | 大概 | 中性的 | 容忍 |
| DKKL1(E214K) | 可能 | 中性的 | 破坏 |
| MPV17L2 (M72V) | 良性 | 有害的 | 容忍 |
| TNFRSF1A (R121Q) | 可能 | 中性的 | 容忍 |
| DKKL1(G187S) | 良性 | 中性的 | 容忍 |
| SCO2(R20P) | 良性 | 中性的 | 容忍 |
| MANBA (T701M) | 良性 | 中性的 | 容忍 |
| MMEL1 (M518T) | 良性 | 中性的 | 容忍 |
| CLEC2D (L23V) | 良性 | 中性的 | 容忍 |
| IL7R (T244I) | 良性 | 中性的 | 容忍 |
| FCRL3(N28D) | 良性 | 中性的 | 容忍 |
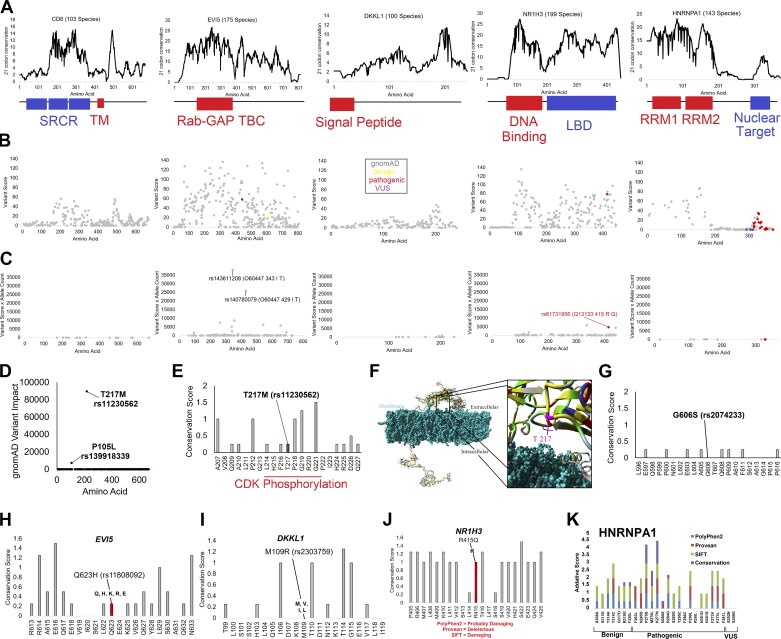
图 3.来自全基因组关联研究 (GWAS) 和 ClinVar 的多发性硬化症 CD6、EVI5、DKKL1、NR1H3 和 HNRNPA1 错义变异。A :对每个基因 (CD6、EVI5、DKKL1、NR1H3、HNRNPA1) 进行的深度密码子进化分析,物种数量显示在括号中,注释域显示在下方。B :来自 ClinVar(良性 = 黄色、致病 = 红色、意义不明确的变异 VUS = 洋红色)和 gnomAD(灰色)的所有变异的排名。C :使用 gnomAD 等位基因计数按变异得分对仅位于保守或选择位点的变异进行综合影响。D –G:CD6,右上角所有 gnomAD 变异对 CD6 的相对预测影响,标识出 T217M 的影响最大 ( D )。E:位点 217 附近的氨基酸保守,该位点位于潜在磷酸化位点的中间。F :嵌入脂质膜(青色)内的 CD6 模型,其保守性用 ConSurf 着色(黄色 = 疏水性保守,蓝色 = 碱性保守,红色 = 酸性保守,绿色 = 极性保守,灰色 = 不保守),右侧为位点 217 的放大视图。G:CD6 的另一个连锁不平衡 (LD) 错义单核苷酸多态性 (SNP),在氨基酸 606 附近显示无或弱保守性。H : EVI5 ,位点 623 周围的氨基酸保守,在整个进化过程中发现多种氨基酸。I :DKKL1,氨基酸 109 周围的保守性,没有保守性。J :NR1H3,位点 415的ClinVar 基因突变序列,位于蛋白质的高度保守区域。K:HNRNPA1,使用多种工具和多发性硬化症基因检测项目组的保守分析评估该基因的所有 ClinVar 基因突变序列。
对于这五种蛋白质中的每一种,多发性硬化症基因检测项目组都在其基因中确定了高度保守和密码子选择的关键位点(图3A),结合 PolyPhen2、Provean 和 SIFT 对 ClinVar 和 gnomAD 工具中发现的每种变异的预测 (图3B),代表了超过 130,000 个已测序个体。基于使用多种工具进行的保守性分析,所有五个基因内的变异都已预测了功能结果 (图3B),其中大多数变异都出现在罕见频率水平,这表明在将等位基因计数纳入指标时,得分没有提高(图3C)。EVI5 有两个最常见的潜在破坏性变异,rs143611208 (I343T) 和 rs140780079 (I429T),但迄今为止尚未对这些变异进行研究,很可能是由于它们在非芬兰高加索人中的等位基因频率较低(rs143611208 在 0.1% 高加索人中,其中阿什肯纳兹犹太人最高为 1.8%,rs140780079 在 0% 高加索人中,其中东亚人最高为 1.6%)。这两个变异在进化上都是 100% 保守的,并且在所有使用的工具中都有破坏性预测。NR1H3 (R415Q) 中的 ClinVar 致病注释变异 rs61731956 是唯一已知的与所有五个基因都有等位基因计数的疾病相关变异,并且有强有力的保守性证据(图3C)。
深入研究这五个基因的疾病相关错义变异(图 3, D – K ) 多发性硬化症基因检测项目组首先对 CD6 进行评估。CD6 T217M (rs11230562) 的综合得分为 89,753,这源于 PolyPhen2 和 SIFT 中的破坏性预测、高于平均值的 −0.6 的 dN-dS 值、位点周围氨基酸的高度保守性以及苏氨酸上的潜在 CDK 磷酸化位点 (图3E)。43% 的阿什肯纳兹犹太人存在该变异,与多发性硬化症(MS)的 rs11230562 领先 SNP 的 R 2相关性为 0.943。CD6 上的 T217 位点预计暴露在表面,靠近多个其他保守氨基酸(图3F)。与 rs11230562 存在 LD 的另一个潜在功能性错义基因突变序列是 rs2074233 (CD6 G60S),但该位点在该区域没有保守性或选择性(图3G)。
另外两个来自 LD SNPs 的错义基因突变序列具有多个破坏性预测,EVI5 Q623H(图3H) 和 DKKL1 M109R (图3),保守性较低,周围保守氨基酸很少。NR1H3 R415Q 基因突变序列的多发性硬化症(MS)关联最近受到争议 ( 45 ),但预测该基因突变序列在 PolyPhen2、Provean 和 SIFT 中是有害的,在多发性硬化症基因检测项目组分析的 NR1H3 的 199 个物种序列中 100% 保守,并且附近有许多其他氨基酸具有高度保守性 (图3J),表明该变异具有功能性。HNRNPA1 与多发性硬化症(MS)自身抗原相关,ClinVar 包含注释为良性、致病性和意义不明确的变异,其中变异 P275S 和 K277N 具有最高的功能预测(图3K)。多发性硬化症基因检测项目组对每个基因的保守性分析可以在多发性硬化症基因检测项目组的 figshare 页面上找到,从而可以对其他人/未来工作中发现的其他变异位点进行额外分析。从高度保守区域的突变导致多发性硬化症(MS)表型这一基本假设开始,这将表明 GWAS 中 LD 区块内的其他非编码变异与多发性硬化症(MS)关联的大多数 LD 区块有关,而不是错义变异。
MS 基因表达
分析多发性硬化症(MS)相关基因组区域的 LD 区块中的错义变异几乎无法识别导致病理的蛋白质通路和细胞类型。因此,多发性硬化症基因检测项目组接下来使用多个表达数据库来研究来自表1的富含多发性硬化症(MS)病理的功能细胞类型。使用人类 FANTOM 数据库,多发性硬化症基因检测项目组绘制了 1,829 个样本中每个基因的表达情况。利用每个组织中每个基因的 z 分数,绘制出多发性硬化症(MS)基因组织特异性表达最多的组织,结果显示大多数组织(99.9%,1827/1829)的多发性硬化症(MS)相关基因的平均 Z 分数小于 1,同时 z 分数大于 2 的单个基因也较少(图4A),即在所有组织中高于平均值两个标准差的值。MS 基因表达的极端值包括多个神经区域(黑质、枕叶皮质和颞叶内侧回)和免疫细胞(单核细胞衍生的巨噬细胞和中性粒细胞),这与已知的多发性硬化症(MS)免疫神经交集一致。在前两个组织特异性数据集(颞叶内侧回和中性粒细胞)中对单个基因的 z 分数进行细分,发现多发性硬化症基因检测项目组列表中的大多数基因都是免疫系统或神经元所特有的(图4B)。只有一个基因,PRDM2,对两种组织都具有高度特异性,这表明存在独特的重叠。
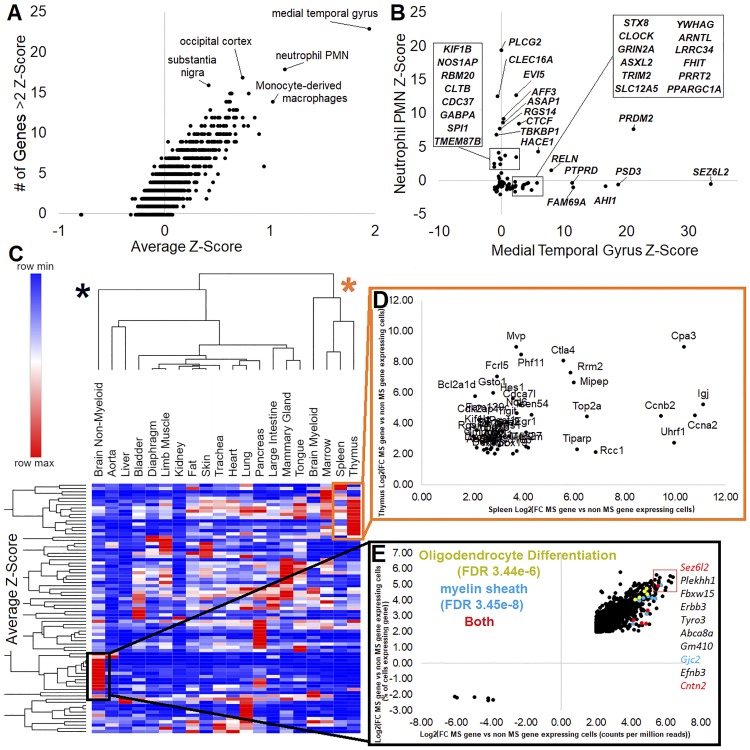
图 4.多发性硬化症列表中的人类基因表达。A :人类 FANTOM 数据库中组织的 z 分数度量,表示该组织中 z 分数 >2 的多发性硬化症 (MS) 相关基因的数量(y轴,即表达水平至少比平均值高两个标准差)和组织中多发性硬化症(MS)基因的平均 z 分数(x轴)。映射到右上方的基因被标记为可能是多发性硬化症(MS)基因表达最高的组织。B :中性粒细胞( y轴)和内侧颞叶(x轴)中候选基因的表达 z 分数,分离免疫系统和神经元的基因。C :每个多发性硬化症(MS)相关基因平均表达的热图(表1) 遍布 20 个单细胞器官数据集。黑色框中的基因主要与非髓系脑细胞有关,橙色框中的基因与免疫系统有关。D :胸腺 ( y轴) 或脾脏 ( x轴) 中表达或不表达多发性硬化症(MS)基因的细胞的基因聚类相关性。E :脑非髓系数据集中表达富集的基因 ( y轴) 或与多发性硬化症(MS)基因相似的细胞数量 ( x轴)。
从中性粒细胞表达中鉴定出的与多发性硬化症(MS)相关的基因包括:PLCG2,小鼠敲除导致中性粒细胞募集减少,人类突变与自身炎症性疾病和 HLA II 类通路改变有关;CLEC16A与多种自身免疫性疾病有关,如 MS、系统性红斑狼疮和银屑病;EVI5,异位病毒整合位点 5 基因,已鉴定出与多发性硬化症(MS)相关的 OR 高达 2,但其机制尚不明确;AFF3与多种自身免疫性疾病有关,包括关节炎、狼疮和 1 型糖尿病;ASAP1是一种肌动蛋白调节剂,与细胞运动和转移密切相关;RGS14与微管相关,参与细胞增殖、信号传导,在小鼠 EAE 模型中上调;TBKBP1参与脊柱炎 和 TNF-α 信号转导。
从内侧颞回表达中鉴定出的与多发性硬化症(MS)相关的基因包括:SEZ6L2,它是最富集的神经基因,此前已发现与神经突生长、ASD和癫痫有关;PSD3虽然对大脑有高度特异性,但在神经细胞中的作用鲜为人知;AHI1已被证明与抑郁症和 Joubert 综合征(一种运动神经发育障碍)有关;FAM69A ( DIPK1A ) 在内质网中发挥作用并与多种神经系统疾病有关;PTPRD是与许多疾病、dota2吧雷电竞 和药物成瘾相关的变异,但很少有研究确定其在神经元中的作用;RELN,也称为 reelin ,双重突变会导致无脑畸形,即大脑光滑,并被认为与神经肌肉连接以及 VLDLR 和脂质生物学有关;昼夜节律基因CLOCK和ARNTL具有与多发性硬化症(MS)相关的 SNP,并且在 EAE 小鼠中表达发生改变。
为了将多发性硬化症基因检测项目组的基因列表扩展到多发性硬化症(MS)功能细胞类型的其他预测网络中,多发性硬化症基因检测项目组利用来自 20 个小鼠器官的单细胞 RNA-Seq 数据集来识别具有共表达基因网络的多发性硬化症(MS)基因富集细胞(图4C)组织聚集显示免疫组织中几种基因的表达升高(橙色框,图4D)和脑非髓系细胞(黑框,图4E)。脾脏和胸腺中细胞的表达量最高,仅表达少量的多发性硬化症(MS)基因。对表达多发性硬化症(MS)相关基因的脾脏和胸腺单细胞与不表达多发性硬化症(MS)基因的细胞进行筛选,发现另外 35 个基因(Cpa3、Ccna2、Ccnb2、Igj、Mipep、Uhrf1、Phf11、Ctla4、Fcrl5、Top2a、Rrm2、Tsen54、Cdca7l、Psat1、Gsto1、Rcc1、Nol6、Tigit、Tfb2m、Fam129c、Cd160、Bcl2a1d、Slc29a1、Tfdp1、Med27、Aarsd1、Gmppb、Ddx51、Serpine2、Cox19、Chpf、Ide、Lcmt1、Rnmt和Dkc1)在两种组织中重叠表达(图4D)。在这些数据中识别出Igj是值得注意的,因为它在多发性硬化症(MS)患者中升高。这 35 个基因在相互作用中具有显著富集(FDR 0.0002),并且根据 GO 富集和字符串网络分析,在有丝分裂细胞周期转换中富集(FDR 3.57e-2),更具体地说是在 G0 和早期 G1 中富集(FDR 3.26e-2)。对大脑的非髓系细胞进行分层,发现有 1,709 个基因与多发性硬化症(MS)相关基因共表达(图4E)。在这 1,709 个基因中,少突胶质细胞分化本体论 (FDR 3.44e-6) 和髓鞘基因 (FDR 3.45e-8) 显著富集。因此,表达多发性硬化症(MS)相关基因的大脑非髓系细胞是参与神经元髓鞘形成的少突胶质细胞。有趣的是,之前很少有相关基因被注释为具有这种生物学作用,这表明单细胞的分离策略确定了可能参与多发性硬化症(MS)生物学的新基因。Sez6l2在多发性硬化症基因检测项目组的多发性硬化症(MS)基因列表和大脑分离列表中,是大脑单细胞数据中相关性最高的基因之一,但之前从未与少突胶质细胞联系起来。第二个小鼠单细胞数据库 Mouse Cell Atlas (MCA)也表明Sez6l2在髓鞘少突胶质细胞中表达升高(P值为 5.8e-155),进一步验证了该基因的这一新作用。分离列表顶部的其他基因包括:Galnt6,大量参与髓鞘维持所需的高尔基体 O-连接糖基化;Plekhh1在所有细胞中研究得很少;Erbb3已知参与少突胶质细胞成熟;Tyro3已显示参与施万细胞髓鞘形成;Efnb3与多发性硬化症(MS)中的髓鞘再生有关。根据 MCA,发现所有基因在髓鞘少突胶质细胞中均高表达。多发性硬化症基因的综合网络从系统层面丰富了髓鞘形成和少突胶质细胞生物学,为未来工作中的进一步网络推断和测试提供了可能。
非编码基因突变序列
从基因表达转向可能影响表达的变异是一种开始过滤多发性硬化症(MS)LD 区块内非编码变异的方法。除了上述少数错义变异外,MS 相关 LD 区块中的大多数变异尚未评估其功能结果(图 3)。为了进一步分析 GWAS LD 区块的非编码基因突变序列,多发性硬化症基因检测项目组使用了 GTEx eQTL 数据库和 RegulomeDB 工具(表1)。eQTL 可以告诉多发性硬化症基因检测项目组 LD 阻断的任何非编码变异是否与基因表达变化相关,而 RegulomeDB 等工具可以根据转录因子结合和表观基因组学见解缩小潜在的因果变异范围。
对于多发性硬化症(MS)列表中的基因,多发性硬化症基因检测项目组发现脂肪组织中的 eQTL 最多(30 个基因 = 33.8%),其次是脑组织(图5A)。从全基因组来看,脂肪组织确实含有最多的 eQTL,因此从多发性硬化症(MS)网络中识别它并不奇怪。将这些值相对于 GTEx 中观察到的具有 eQTL 的基因总数进行标准化,会导致组织具有相对均匀的值(图5A,红色)。其中一些基因的 eQTL 存在于多种组织中(图5B),即EVI5 (23 种组织 = 54.8%)、HLA-DRB1 (22 种组织 = 52.4%)、HLA-DRB5 (21 种组织 = 50%) 和MERTK (20 种组织 = 47.6%)。处理多发性硬化症(MS)相关基因在脂肪组织中的效应大小与显著性时,发现所有相关基因在 LD 中都有多个 eQTL。MERTK 对基因表达增加具有最显著的遗传影响 (图5C)。MERTK是一种受体激酶,已知其罕见和常见基因突变序列与多发性硬化症(MS)有关,并且与蛋白质伴侣有关,这些蛋白质伴侣显著丰富了胰岛素和脂肪酸生物合成成分的 KEGG 通路(相对 FDR 0.005 和 0.013)。MERTK 表达的非编码基因突变序列在脂肪中的平均效应大小为 9.45,在胫骨神经元中的平均效应大小为 11,这表明MERTK可能具有影响神经元脂肪酸合成过程和髓鞘形成动力学的非编码基因突变序列。
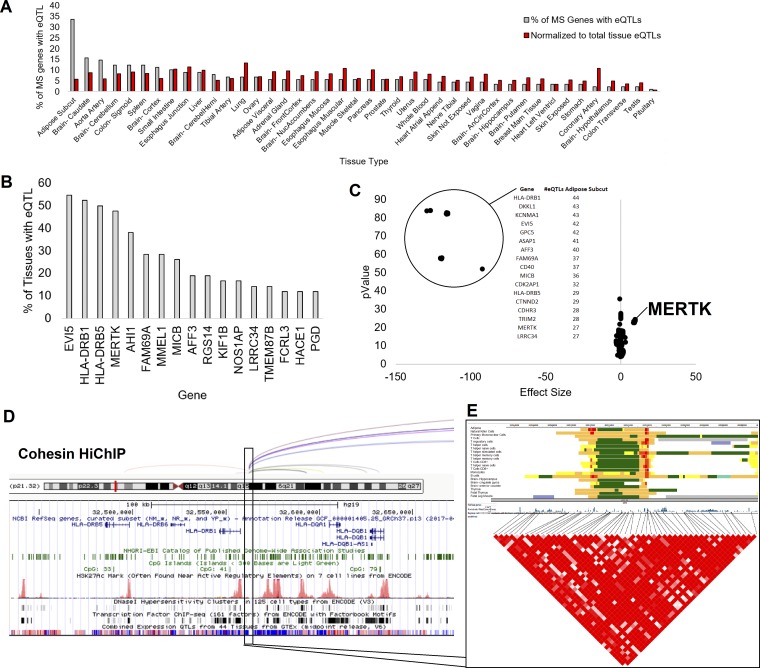
图 5.多发性硬化症的表达数量性状基因座 (eQTL) 分析全基因组关联研究 (GWAS) 连锁不平衡 (LD) 单核苷酸多态性 (SNP) 以提名基因和表观遗传机制。A :列表中多发性硬化症(MS)基因的百分比,其中多种人体组织中存在 eQTL(灰色),以及相对于每种组织内映射的 eQTL 总数(包括非多发性硬化症(MS)相关基因)的标准化值(红色)。B :具有 eQTL 的组织最多的基因。C :皮下脂肪中 eQTL 的效应大小(x轴)和P值(y轴),突出显示效应大小最大的基因。D :HLA-DRB1/5 区域附近的功能性 SNP 视图。顶部显示的是该位点与远端区域相互作用的 Cohesin Hi-ChIP 循环数据。E :几种组织的 ChromHMM 模型,显示该区域是潜在的增强子。下面显示的是 LD SNP 相关性。
脂肪中最大的效应量是 HLA 基因HLA-DRB1/DRB5的效应量。HLA 风险基因相互作用与肥胖在多发性硬化症(MS)发展中的高度关联表明 eQTL 的重要性值得关注。根据 Cohesin HiChip 数据集,这些调控 eQTL 的 LD 区块循环回 HLA-DRB1 和 HLA-DRB5 的启动子(图5D)。LD 区块中有一小部分区域被标注为功能性,这是基于 Roadmap Epigenomics 在多种细胞类型(包括脂肪细胞、自然杀伤细胞、多种 T 细胞类型、单核细胞、B 细胞、胸腺、脑区和腿部肌肉)中标注的状态(图5E)。23 个基因突变序列与 HLA-DRB1 和 HLA-DRB5 的领先 SNP 具有 0.8 R 2相关性。人们对调节该区域的 TF 知之甚少,突出表现为缺乏 ENCODE TF 结合位点映射和 RegulomeDB 的分数(rs9271683 和 rs9271689 的分数为 3a)。
通过 RegulomeDB 对所有 LD SNP 进行进一步分析,发现了几个预计会发生改变且可能对多发性硬化症(MS)病因很重要的 TF 结合位点(表格1)这些变异之一 rs61784580 与主要 SNP rs10492972 具有 0.8 R2相关性,与多发性硬化症(MS)的几率比为 1.34 相关,并预计会改变 GFI1 的结合位点(图 6)。领先的 SNP 位于KIF1B基因的内含子内,是KIF1B表达的 eQTL (图 6,黑框),落在没有已知 ENCODE TFs 结合的位点上,预计不会改变任何 TF 结合位点。然而,rs61784580 位于距离主要 SNP 82,616 个碱基对处,位于 H3K27 酰化(活性转录标记)Roadmap 注释的启动子上,该启动子附近有 26 个来自 ENCODE 的 TFs 结合,环路到多个基于黏连蛋白 HiChIP 的 Roadmap 注释增强子,是KIF1B eQTL,预计会改变 GFI1 TF 结合位点(图 6,青色框)。GFI1 是一种对髓系和淋巴细胞发育至关重要的基因,其突变与中性粒细胞减少症和 T 细胞发育有关。因此,仅从数据预测来看,多发性硬化症基因检测项目组认为 rs61784580 在多种细胞类型中调节KIF1B的机制性非编码生物学与多发性硬化症(MS)相关,应在实验室中进一步研究。
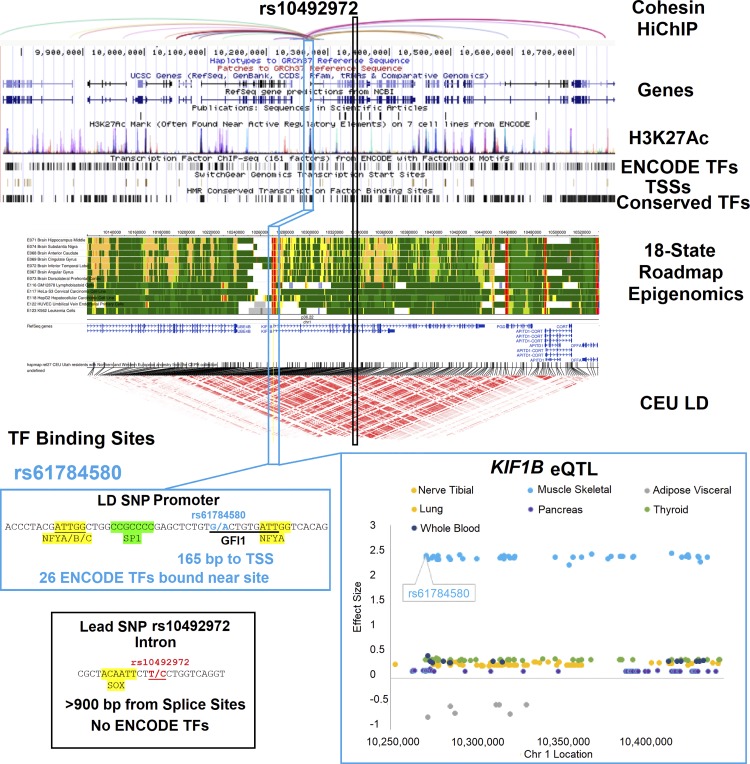
图 6:rs10492972 区域中功能性非编码 SNP 的 RegulomeDB。从顶部开始是rs61784580 启动子 SNP 或内含子引导 SNP rs10492972 的循环数据,后面是人类基因组浏览器的多个数据集、该区域的 18 状态 ChromHMM 基因组注释、CEU LD SNP 相关性、两个位点附近转录因子结合位点的详细信息以及来自 LD 块的 KIF1B 的 eQTL,它们都显示出相似的效应大小。
结合数据来剖析多发性硬化症(MS)LD 生物学
MS 的 GWAS SNP 与 GWAS 目录中所有其他特征的交集可用于解释疾病病理的重叠机制和导致病理的特征。根据 GWAS 数据库查询了多发性硬化症(MS)主导 SNP 的整个估算 SNP 数据集,删除了多发性硬化症(MS)相关特征,揭示了重叠遗传学与多种其他自身免疫性疾病(如克罗恩病、炎症性肠病、狼疮、乳糜泻、关节炎和糖尿病)以及罕见的神经系统疾病多系统萎缩(图7A)。一些基因突变序列被发现具有多种相关特征,包括一些 LD 基因突变序列(图7B)。对这些具有多种性状的SNP的剖析阐明了本文结合数据整合的多个层次的知识(图7C),凸显了多发性硬化症基因检测项目组目前的研究在将机制见解映射到多发性硬化症(MS)系统生物学方面的能力。总结图7C对于多发性硬化症(MS)相关非编码变异的顶级调控预测:1 ) 发现 rs140522 的 LD 阻断与多种血液性状和细胞类型相关,影响巨噬细胞中的TYMP表达;2 ) 发现 rs10936599 的 LD 阻断与多种癌症和免疫功能障碍相关,其基因突变序列可能会改变 STAT5 结合,影响室管膜神经细胞中的LRRC34表达;3 ) rs1036207 的 LD 阻断与嗜酸性粒细胞和中性粒细胞生物学相关,其基因突变序列可能会改变 Evi-1 转录因子结合,影响多种细胞类型中的 NDFIP1 表达。
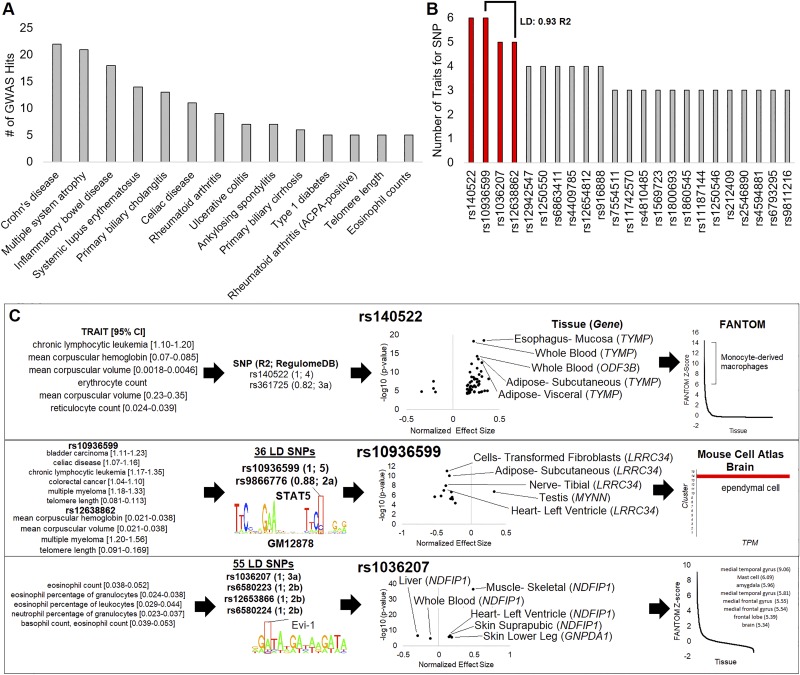
图 7:多发性硬化症 (MS) 连锁不平衡 (LD) 单核苷酸多态性 (SNP) 与其他全基因组关联研究 (GWAS) 性状重叠。A :每个性状映射的与任何多发性硬化症(MS)相关 LD 区段基因突变序列重叠的 SNP 数量。性状按发生情况排序。B :与其关联性状最多的 SNP,这些 SNP 也位于多发性硬化症(MS)LD 区段内。前四个在C中详细说明,其中两个 SNP 位于 LD,相关性为 0.93R 2。C :性状的三个前 SNP细分。对于每一个,最左边显示具有 95% 置信区间的性状。接下来是通过 RegulomeDB 对功能性 LD SNP 的分析,显示每个 SNP 相对于领先 SNP 的 R2 值和基因突变序列的 RegulomeDB 得分。此后是顶级 RegulomeDB SNP 的 eQTL 列表。最右边显示了来自 FANTOM(免疫细胞)或 Mouse Cell Atlas(大脑)的具有最显著 eQTL 的基因的表达数据。
多发性硬化症基因检测科学性及其位点选择
遗传因素对疾病和特征的影响已得到充分证实,但人们对变异发挥作用的机制和途径了解甚少,无法了解其遗传关联。缺乏机制的原因有几个,最明显的是多个变异的 LD 遗传,因此很难从统计学上找出致病变异。为了解决这个问题,人们在表观遗传学和 TF 结合方面投入了大量资金,但工具集成到疾病病理学解释方面却存在滞后。虽然目前的数据和计算能力很强,但理解数据联系仍然需要人工时间来整理信息并解释遗传因素对疾病的影响。多发性硬化症基因检测项目组在致病基因鉴定方面证明了这一点,展示了如何需要多种工具来识别 LD 变异对疾病的贡献。利用数据库和工具集成的系统遗传策略来推导可能的多发性硬化症(MS)病因的相关网络。这些数据表明了多个部位的潜在机制,突出了免疫系统成分和神经髓鞘生物学的交集(图 8)。虽然可以使用现有工具快速筛选错义变异,但非编码变异的解释在数据整合方面仍然具有挑战性,但多发性硬化症基因检测项目组可以确定一些功能位点,正如《多发性硬化症基因检测》所讨论的那样。许多其他遗传学尚未通过现有工具得到很好的理解,这表明需要针对巨噬细胞和 T 细胞转录控制开关定制的额外数据集,这些开关可能会因非编码变异而改变。自 1868 年最初将多发性硬化症(MS)描述为一种独特疾病以来,对该疾病遗传机制的解释仅在过去十年中才出现。多发性硬化症基因检测项目组现在对基因序列变化对多发性硬化症(MS)的贡献的了解程度是惊人的,这是数百万美元投资于遗传关联的结果,更重要的是,这是更大的一般转录数据集的结果。随着这些数据集变得越来越便宜和更容易生成,应该开发更多定制的数据集以完全解释多发性硬化症(MS)遗传学,为风险倾向和个性化治疗建立更强大的计算算法。
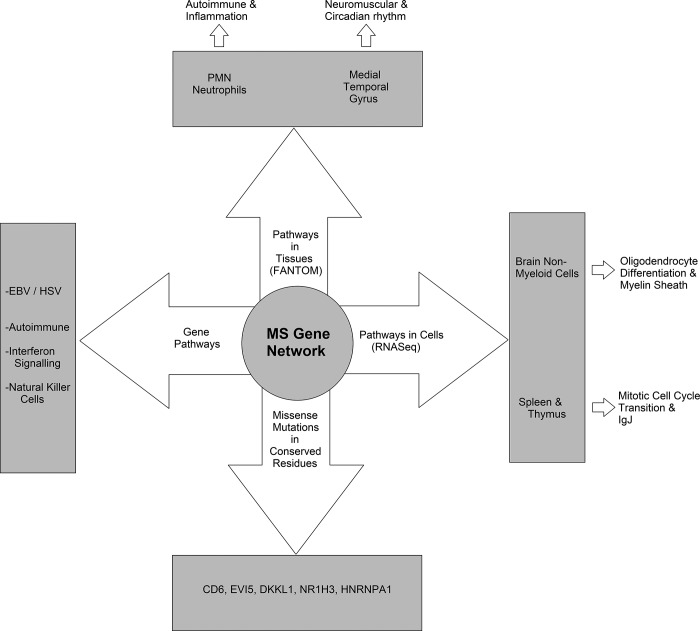
图 8:基于多发性硬化症基因检测项目组的遗传系统数据整合的与多发性硬化症 (MS) 病因相关的途径的工作模型。
(责任编辑:佳学基因)









































